Membranproteine Wie kommt der Zucker in die Zelle?
Membranproteine sind essenziell für die Kommunikation der Zelle mit ihrer Umgebung und sorgen für den Transport wichtiger Stoffe über die Zellmembran. Ihre besonderen Eigenschaften machen diese Proteine zum geeigeneten Ansatzpunkt für Medikamente. Auf der Grundlage eines neuen bioinformatischen Ansatzes gelang es einem internationalen Forscherteam nun, die Struktur eines wichtigen Zuckertransporters aufzuklären.
Anbieter zum Thema
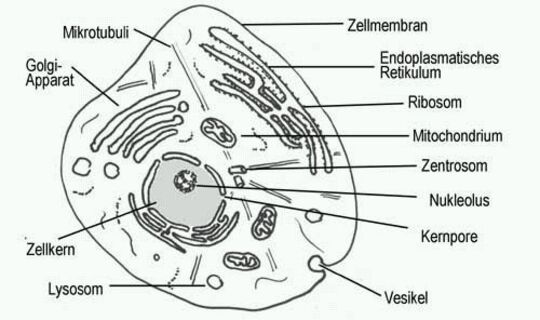
München, New York/USA – Im Rahmen einer groß angelegten Kooperation ist es Wissenschaftlern der amerikanischen Columbia University gelungen, die dreidimensionale Struktur des bakteriellen Membranproteins ChbC mittels Kristallstrukturanalyse zu bestimmen. Die Proteinfamilie, zu der ChbC gehört, ist essentiell für den Transport spezifischer Zucker in die Zelle und stellt damit einen wesentlichen Faktor für das Überleben des Bakteriums dar. Wie diese Proteine jedoch den Zucker in die Zelle schleusen, war bisher nicht bekannt.
Möglich machten dieses Ergebnis Vorarbeiten des New York Consortium on Membrane Protein Structure (NYCOMPS). Im besonderen Fokus der Wissenschaftler stehen Membranproteine. „Jedes vierte Protein ist ein Membranprotein und doch kennen wir nur von etwa fünf Prozent von ihnen die Struktur. Hier stehen wir noch vor einer großen Herausforderung“, so Burkard Rost, Professor
für Bioinformatik an der TU München. Da die experimentelle Analyse eines Membranproteins mehrere Jahre dauern kann, kommt der Auswahl vielversprechender Kandidaten für die aufwändige Analyse eine große Bedeutung zu., Der Pool potentiell interessanter Sequenzen ist groß. Über 300.000 Proteinsequenzen aus den Genomen von 96 Organismen standen zu Beginn des Projektes zur Auswahl. Nur ein Bruchteil dieser Sequenzen kann jedoch überhaupt experimentell analysiert werden.
Gemeinsam mit anderen NYCOMPS-Bioinformatikern identifizierte Burkhard Rost, ChbC als lohnendes Ziel. Ausgangspunkt der NYCOMPS-Wissenschaftler ist eine bioinformatische Methode aus dem Gebiet der „Strukturgenomik“ (engl. „Structural Genomics“). Die grundlegende Annahme dieser Methode ist, dass Proteine mit gemeinsamen evolutionären Vorfahren, sogenannte „Proteinfamilien“, sich sowohl in ihren Aminosäuresequenzen als auch in ihrer dreidimensionalen Struktur gleichen. Ist die Struktur eines dieser verwandten Proteine durch Experimente bekannt, lassen sich auch die restlichen vorhersagen.
Bioinformatischer Ansatz führt zum Ziel
Um ein geeignetes Protein zu finden, entwickelten die NYCOMPS-Wissenschaftler einen gängigen bioinformatischen Ansatz weiter. Statt wie üblicherweise zu Beginn des Projekts alle Proteine in einer Genom-übergreifenden Karte anzuordnen und dann eine Auswahl zu treffen, bauten die Bioinformatiker die Proteinfamilien jeweils um eine einzelne Sequenz herum auf. Der Vorteil eines solchen „Kernproteins“ („seed protein“) ist, dass die Wissenschaftler interessante Zielsequenzen sofort zu Proteinfamilien ausweiten können, statt diese zuerst in der bereits bestehenden Proteinkarte suchen zu müssen. Die Wahrscheinlichkeit innerhalb der um das Zielprotein herum kreierten Familie einen für die späteren Experimente geeigneten Verwandten zu finden ist ebenfalls höher.
Als Zielproteine filterten die NYCOMPS-Wissenschaftler schrittweise die Proteine heraus, die Eigenschaften von Membranporteinen aufwiesen. Dann erweiterten sie diese zu Proteinfamilien und wählten wiederum diejenigen Sequenzen, die für die Experimente besonders geeignet waren. All das geschah rein virtuell durch mathematische Vorhersagen und ganz ohne Experimente. Im Fall von ChbC blieben letztlich aus der Vielzahl an Sequenzen 25 aus 13 Organismen übrig. Erst diese 25 wurden dann im Labor näher untersucht. Letztlich führte eine der Sequenzen zum Erfolg - die sprichwörtliche „Nadel im Heuhaufen“ war gefunden.
(ID:26626510)





:quality(80)/p7i.vogel.de/wcms/aa/06/aa065b925f536c86d1de68e56f85e5e7/0128901830v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/73/36/733639105fa0d1883703f681d36029e5/0128891779v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/9a/2c/9a2c29461a7363766921da706c18915a/0129780133v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/77/05/7705ae165791fa98030b6276a92edbdc/0129450629v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/d6/a7/d6a793a55f871890d809605bfca2bf13/0128913698v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/f2/3e/f23e822277e907f2f56360ab3247cbe0/0129330424v1.jpeg)
:quality(80)/p7i.vogel.de/wcms/8e/0c/8e0c352eb77a74164d8d4b218f08ada7/0129146123v1.jpeg)
:quality(80)/p7i.vogel.de/wcms/b1/15/b1150724d5d5d05a0cc964e35da83967/0129822559v1.jpeg)
:quality(80)/p7i.vogel.de/wcms/af/8c/af8c3e6b6a4c3aaf978efca0598dd91f/0129745187v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/81/48/81487a3895f2531f491f3d21095e21f7/0129745376v1.jpeg)
:quality(80)/p7i.vogel.de/wcms/34/fe/34fec2ab6938e1f86fe8eeffef1960c1/0129649873v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/f3/ff/f3ff704238770849c8ea6501e1875c84/0129639452v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/cf/12/cf12c9cb9f9cbb200c85faab6ca37589/0129611307v2.jpeg)
![Abb. 1: Mehr als 150 Milliarden Mikroplastikpartikel gelangen jedes Jahr allein aus einer mittelgroßen kommunalen Kläranlage in unsere Gewässer [1]. (Symbolbild) (Bild: © Thirawat - stock.adobe.com) Abb. 1: Mehr als 150 Milliarden Mikroplastikpartikel gelangen jedes Jahr allein aus einer mittelgroßen kommunalen Kläranlage in unsere Gewässer [1]. (Symbolbild) (Bild: © Thirawat - stock.adobe.com)](https://cdn1.vogel.de/Ig0DZk804N0u6g0JY0IDnFhcGWs=/288x162/smart/filters:format(jpg):quality(80)/p7i.vogel.de/wcms/e9/bf/e9bf6aa58a1c17d9381091925a75d7bc/0128824528v1.jpeg)
:quality(80)/p7i.vogel.de/wcms/df/da/dfda1c2bee3543f7b2df8833859c8b09/0129818894v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/2e/17/2e1795b6750cc3eaad6ba81c0bc79d16/0100811496v7.jpeg)
:fill(fff,0)/p7i.vogel.de/companies/66/71/66716b11ad4e1/gerstel-logo-mlw.png)
:fill(fff,0)/images.vogel.de/vogelonline/companyimg/15200/15260/65.jpg)
:fill(fff,0)/images.vogel.de/vogelonline/companyimg/84700/84716/65.jpg)
:quality(80)/p7i.vogel.de/wcms/b0/8f/b08fa449df88dd30a2d3a51fc8da23af/0123785376v2.jpeg)
:quality(80)/p7i.vogel.de/wcms/8c/d0/8cd09f96a01e4c80aded0d19512923d0/0126565309v2.jpeg)